# Pourquoi la métagénomique?
### Ecosystèmes microbiens
- Majeure partie de la biomasse sur Terre
- 90% des cellules d'un être humain
- Colonisation de la plupart des niches écologiques
### Métagénomique
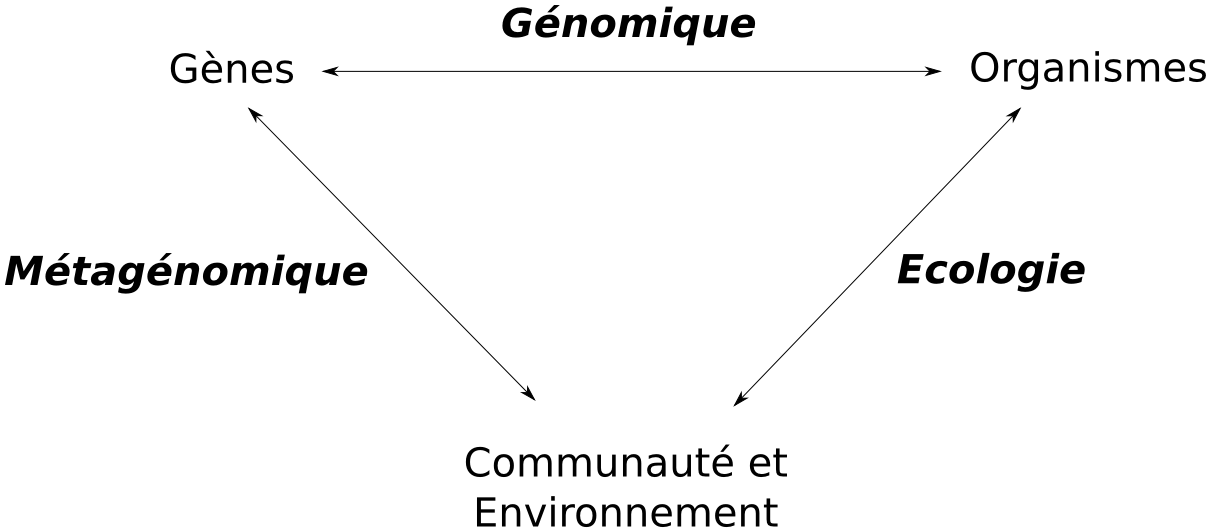
### Métagénomique
Compréhension du rôle écologique, du métabolisme et de
l'histoire évolution d'un cosystème
### Métagénomique
- Pas de culture nécessaire
- Identification pour un écosystème
- Micro-organismes (composition)
- Gènes
- Fonctions métaboliques
### Projets métagénomiques
- 2 165 [projets sur NCBI](http://www.ncbi.nlm.nih.gov/bioproject/?term=metagenomic) (Janvier 2016)
- Sable de plage
- Moustique
- Corail
- Glace
- Air de la ville de Singapour
- Surface de la cuvette des toilettes
- Fromages
- ...
# Principes de la métagénomique
### Métagénomique
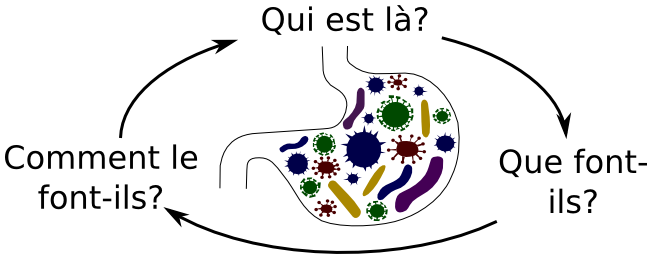
### Métagénomique
Objectifs:
- Identifier les organismes présents dans un échantillon
- Identifier les fonctions principales réalisés par les
organismes d'un échantillon
## Extraction et séquençage
### 2 types d'expériences métagénomiques
- Amplicon
- "Envionmental shotgun metagenomics", WGS ou métagénomique
### "Envionmental shotgun metagenomics", WGS ou métagénomique
Séquençage de l'ensemble des séquences d'un échantillon
### Prétraitements
- Contrôle de la qualité des séquences
- Assemblage
- Tri des séquences
- Prédiction des séquences d'intérêt
### Tri des séquences
- Utilité
- Assignation des séquences
- Recherche dans des bases de données spécifiques
- Caractéristiques d'intérêt
- Gènes ARNr, ARNt
- ORF
- Unités taxonomiques opérationnelles (OTU)
- Quelques outils
- SortMeRNA, rRNA selector
- MetaGeneAnnotator, Prodigal, Orphelia
- tRNAscanSE
## Affiliation taxonomique des séquences
### Pourquoi l'affiliation taxonomique?

### Pourquoi l'affiliation taxonomique?
### Procédure standard
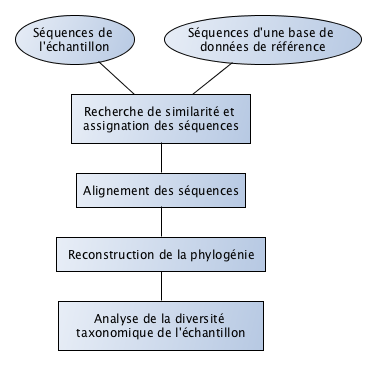
### Limites
- Longueur des séquences
- Nombre de séquences
- Absences d'homologues dans les banques
- Faible abondance des marqueurs phylogénétique classiques
- Boues d'épuration : 0.17%
- Mer des Sargasses : 0.06%
- Sol acide, mines du Minnesota : 0.017%
### Recherche de similarité
- Recherche de similarité par fréquence de k-mer
- Recherche de similarité intrinsèques
- Contenu en GC
- Usage des codons
### Analyse de la diversité d'un échantillon
Alpha diversité
- Notion définie par *Whittaker, Evolution and Measurement
of Species Diversity, 1972*
- Mesure pour un échantillon de
- Richesse
- Nombre de groupes d'individus génétiquement liés
(nombre d'espèces)
- Egalité
- Proportion d'espèces présentes

## Affiliation fonctionnelle
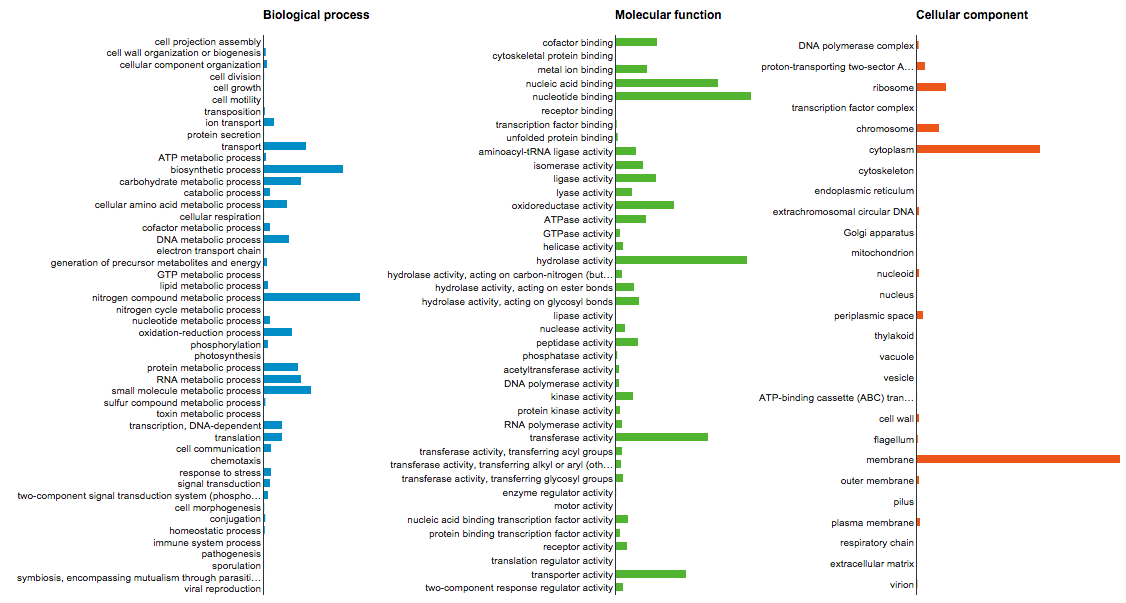
### Pourquoi l'affiliation fonctionnelle ?
### Procédure
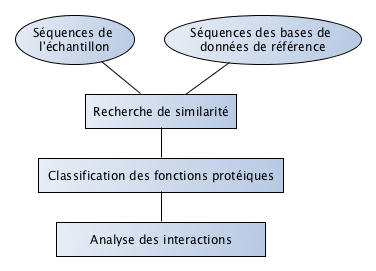
### Classification des fonctions protéiques
- Recherche contre bases de données de références
- Cluster of orthologous groups of proteins (COG)
- SEED
- Kyoto Encyclopedia of Genes and Genomes (KEGG)
- ...
- Recherche de motifs fonctionnels
- InterPro
- Prosite
- ...
## Exemple
[Lean human gut](https://www.ebi.ac.uk/metagenomics/projects/SRP000319/samples/SRS000998/runs/SRR029687/results/versions/1.0#ui-id-6)
## Importance des métadonnées
### Que sont les métadonnées?
Description en profondeur et contrôlée de l'échantillon
- Où?
- Latitude et longitude
- Profondeur
- ...
- Quand?
- Quoi?
- Comment?
- Processus de conservation
- Méthode d'extraction
- Plateforme de séquençage
- ...
### Importance des métadonnées
Indispensable pour partager et intégrer les données dans
la communauté
Besoin d'un vocabulaire standardisé minimal
### Genome Standard Consortium (GSC)
Organisme international, ouvert, formé en 2005
Objectifs
- Promouvoir les mécanismes de standardisation de la
description des génomes
- Promouvoir l'échange et l'intégration des données
génomiques
### Genome Standard Consortium (GSC)
Missions
- Développement
- de nouveaux standards (méta)génomiques
- des méthodes de capture et d'échange des métadonnées
- Harmonisation de la collection des métadonnées et des
efforts d'analyse à travers toute la communauté génomique
### Standards des métadonnées
**MixS** : Minimum Information about any Sequence
**MIGS/MIMS** : Minimum information about a (Meta)Genome Sequence
**MIMARKS** : Minimum Information about a MARKer gene Sequence
# Métagénomique comparative
[*Turnbaugh et al, Nature, 2009*](http://www.nature.com/nature/journal/v457/n7228/full/nature07540.html)
Note:
A core gut microbiome in obese and lean twins
Données pour les TP
## Comparaison de la diversité
## Comparaison des fonctions réalisées
## Conservation des données
### Données
- Besoin d'une profondeur de séquençage importante en
métagénomique
- Augmentation exponentielle des données générées
- Limites des transferts des données par les réseaux
informatiques
Note:
- 2-10 To de données / laboratoire / an
- Plus rapide d'envoyer un disque que de récupérer par
téléchargement?
### Conservation des données
- Quoi?
- Données brutes
- Résultats des analyses
- Description des données
- Où?
- Localement?
- Cloud?
- Dépôts publics?
Note:
- Temps d'analyses peu couteux donc pas besoin de garder
tous les résultats intermédiares
- Importance de la description des données
- Conservation locale
- risque de perte des données
- confidentialité
### Principaux dépôts publics
- EBI : ENA, EBI metagenomics,...
- NCBI
- DDBJ
Note:
- ENA
- métadonnées complets suivants les standards
- outils intuitifs, checklist, support, ...
- NCIB
- metadonnées suivant les standards encouragés
- DDBJ
- pas besoin de suivre les standard GSC
## Traitement des données